
UViT-Seg:一种基于 ViT 和 U-Net 的高效框架,用于在结肠镜检查和 WCE 图像中准确分割结直肠息肉(Python代码实现+完整论文+数据集+UI界面)
U-Net 最初由 Ronneberger 等人 [20] 引入,用于生物医学图像分割,作为各种图像分割任务的首选架构而广受欢迎。这种架构植根于全卷积网络的基础 [48U-Net 模型包括两个关键组件:编码器和解码器路径。编码器的作用是使用一系列具有 3x3 滤波器的卷积层提取深度特征,然后是 ReLU 激活层和 MaxPooling 层。相反,解码器负责创建输出分割映射,采用上采样、卷积、ReL
摘要
结直肠癌 (CRC) 是全球最普遍的癌症之一。内窥镜图像中结直肠息肉的准确定位对于及时检测和切除至关重要,对 CRC 预防有重大贡献。对胃肠道筛查技术生成的图像进行手动分析对医生来说是一项乏味的任务。因此,计算机视觉辅助癌症检测可以作为息肉分割的有效工具。人们已经做出了大量努力来自动化息肉定位,大多数研究依靠卷积神经网络 (CNN) 来学习息肉图像中的特征。尽管 CNN 在息肉分割任务中取得了成功,但由于它们完全依赖于从图像中学习局部特征,因此在精确确定息肉位置和形状方面表现出重大局限性。虽然胃肠道图像的特征表现出显着差异,包括高级和低级特征,但需要一个结合学习息肉两种特征的能力的框架。本文介绍了 UViT-Seg,这是一个专为胃肠道图像中的息肉分割而设计的框架。UViT-Seg 在编码器-解码器架构上运行,采用两种不同的特征提取方法。编码器部分的视觉转换器捕获远程语义信息,而集成挤压激励和双注意力机制的 CNN 模块捕获低级特征,专注于关键图像区域。对包括 CVC clinic、ColonDB、Kvasir-SEG、ETIS LaribDB 和 Kvasir Capsule-SEG 在内的五个公共数据集进行的实验评估证明了 UViT-Seg 在息肉定位方面的有效性。为了确认其泛化性能,该模型在训练中未使用的数据集上进行了测试。以常见的分割方法和最先进的息肉分割方法为基准,所提出的模型产生了有希望的结果。例如,它在 CVC Colon 数据集上实现了 0.915 的平均 Dice 系数和 0.902 的平均交集对和。此外,UViT-Seg 具有高效的优势,用于训练和测试所需的计算资源更少。此功能使其成为实际部署场景的最佳选择。
关键字:结直肠癌、息肉定位、CNN、视觉转换器、注意力机制
1.简介
结直肠癌是最常被诊断出的恶性肿瘤之一,是全球男性和女性癌症相关死亡的主要原因 [1]。美国最近的统计数据将结直肠癌列为导致癌症相关死亡率的第三大因素,估计有 53,020 例新病例和 52,550 例相关死亡。此外,其他消化系统癌症,如食管癌、胃癌和小肠癌,对癌症相关的总死亡率有显著影响 [2]。息肉、溃疡和出血等胃肠道 (GI) 疾病对医生检测和定位小肠内部构成了重大挑战。具体来说,由于息肉的大小、形状和形态特征各不相同,因此检测息肉是一项非常困难的挑战。然而,已经采取了许多举措来改进诊断方法和技术进步,旨在提高息肉检测的准确性和效率。结肠镜检查是用于筛查预防 CRC 癌症的主要工具之一。该手术包括使用经直肠或口腔插入的软管检查大肠。该管配有一个小型摄像头,使医生能够观察结肠内部[3]。在此过程中,专家会寻找息肉并确定是否存在癌症。一般来说,息肉分为两大类:非肿瘤性息肉和肿瘤性息肉。非肿瘤性息肉通常不会发展成癌症,而肿瘤性息肉有可能发展成癌症 [1]。结直肠息肉分为三种不同的大小:小 (5 mm)、小 (6 至 9 mm) 和高级或广泛 (10 mm) [4]。虽然结肠镜检查可以有效地检测和切除息肉,但它确实有几个局限性。这种侵入性手术包括将一根称为结肠镜的软管插入直肠和结肠,这可能会导致不适,可能需要镇静。此外,结肠镜检查可能会漏掉一些息肉,尤其是较小的息肉或位于难以触及区域的息肉 [5]。然而,由于其复杂的结构和大约 6 毫秒的相当长的长度,检查胃肠道内的小肠带来了重大挑战。因此,无线胶囊内窥镜检查 (WCE) 已成为胃肠道的创新诊断工具。WCE 利用摄入的紧凑型 11 毫米胶囊,在人体消化系统中导航,配备一个小型集成摄像头来捕捉胃肠道的图像。值得注意的是,它可以在一次检查中生成大约 55,000 张图像,所有这些图像都传输到外部设备进行全面分析 [6]。WCE 相对于结肠镜检查的主要优势在于其非侵入性,无需工作人员准备。此外,WCE 的非侵入性允许个人在检查期间继续他们的日常活动,提供额外的舒适度 [7, 8]。尽管这些工具增强了胃肠道检查过程,但结肠镜检查和 WCE 数据都需要医生进行人工分析和治疗。对于医生来说,解读大量图像是一项劳动密集型且耗时的任务。因此,迫切需要开发医疗支持系统,以帮助医生做出快速、经济高效且可靠的决定。
利用人工智能 (AI) 系统的计算机辅助检测 (CAD) 的最新发展导致了胃肠道医学图像分析的重大进步 [9, 10]。机器学习 (ML) 和深度学习 (DL) 方法是胃肠道恶性肿瘤分析的主要贡献者。这些技术在提高胃肠道疾病诊断的准确性和效率、帮助医疗保健专业人员做出更明智的决策以及改善患者护理方面显示出巨大的前景。手工制作的方法,如局部二进制模式(LBP)、尺度不变特征变换(SIFT)和定向梯度直方图等,已被用于表征结肠镜检查和WCE图像[11–13]。各种机器学习算法,包括支持向量机(SVM)、随机森林(RF)、K最近邻(KNN)、决策树(DT)等,已被用作分类器来识别经典描述符提取的异常区域特征[14–16]。但是,手工制作的方法在泛化能力方面受到限制,因为它们通常是针对特定任务或数据集定制的。选择合适的 WCE 和结肠镜检查功能的过程在时间和劳动力方面也可能要求很高。
相比之下,基于深度学习的 CNN 在从 GI 图像中学习特征方面表现出有希望的性能 [17–19]。CNN 优于手动方法的优势在于它们能够自动提取相关特征。各种基于 CNN 的编码器-解码器架构,如 U-Net [20]、SegNet [21] 等,已被广泛用于有效定位小息肉,性能令人印象深刻。然而,CNN 有两个明显的缺点:它们需要大量的训练数据,并且可能难以有效地考虑全局信息和空间环境。这些限制会显著影响模型的精度,尤其是在检测和分割方面,因为大多数息肉数据集都包含全局和局部特征的组合,这两者都对于准确识别包含息肉的区域至关重要。另一方面,Vision Transformers (ViTs) 近年来已成为许多计算机视觉任务的领跑者,其性能在 ImageNet 数据集上超过了 CNN。Transformers 已被证明非常有效,因为它们的架构建立在自我注意机制 (SEA) 之上,该机制用于通过考虑所有区域之间的关系来捕获和识别长期依赖关系和全局上下文 [22]。利用 CNN 的能力来获取本地特征并利用 ViT 的潜力来捕获全球背景,可以提供一种增强息肉分割的有效方法。
文献中已经做出了许多努力来改进和推广息肉分割。鉴于息肉数据集中存在许多障碍,实现小息肉的精确识别仍然是一项艰巨的任务。这些障碍包括低对比度、复杂的背景以及形状和纹理的大量变化。如图 1 所示。1、在视觉上区分息肉与周围的健康或正常组织可能具有挑战性,因为它们在颜色和质地方面可能看起来非常相似。因此,迫切需要开发决策支持系统来帮助医生解释 GI 图像中的息肉区域。本文提出了一个基于编码器-解码器架构的深度学习框架,用于息肉分割。编码器建立在 vision transformer 之上,它采用自注意力机制来捕获长期依赖关系并学习全局信息。同时,解码器利用 CNN 的力量与挤压和激发 (SE) 和双注意力机制 (DAM) 相结合,提取局部模式并识别图像中最重要的区域。本研究的主要贡献可以突出如下:
-
提出了一种基于 transformer 编码器和 CNN 解码器的深度学习方法,用于小息肉分割。
-
编码器是使用视觉转换器构建的,该转换器用于从结肠镜检查和 WCE 图像中提取全局模式和空间上下文。
-
解码器部分展示了 CNN 在学习不变局部特征方面的潜力。
-
采用高效的基于双重注意力的残差块来捕获复杂的特征,并增强模型关注空间和通道细节相关信息的能力。
-
squeeze 和 excitation 块战略性地应用于解码器部分的不同阶段,以提高模型在学习特征方面的性能,使模型能够动态强调重要信息。
-
所提出的方法在五个公共数据集上进行训练和验证,每个数据集用于训练,而其他数据集用于测试,反之亦然。
-
模型性能证明了它能够以相当的精度识别息肉区域,优于最先进的息肉分割方法。
-
该模型可用于息肉分割的实际场景,因为它能够在看不见的数据集上泛化和定位息肉。
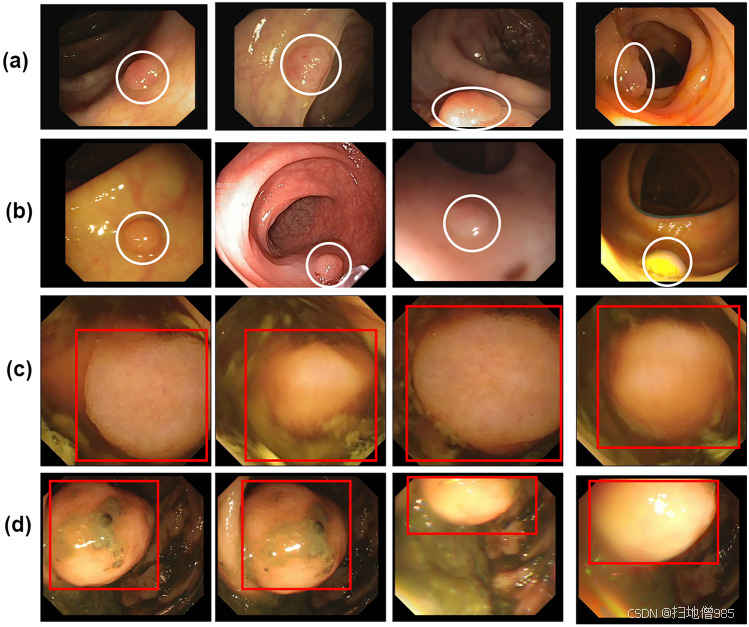
1.息肉的一些示例:前两行 a、b 显示小息肉,而最后两行 c、d 显示大息肉
2.参考文献阅读
2.1References
- 1.Siegel, R.L., Miller, K.D., Wagle, N.S., Jemal, A.: Cancer statistics, 2023. CA: a cancer journal for clinicians 73(1), 17–48 (2023) [DOI] [PubMed]
- 2.Siegel, R.L., Wagle, N.S., Cercek, A., Smith, R.A., Jemal, A.: Colorectal cancer statistics, 2023. CA: a cancer journal for clinicians 73(3), 233–254 (2023) [DOI] [PubMed]
- 3.Hazewinkel, Y., Dekker, E.: Colonoscopy: basic principles and novel techniques. Nature reviews Gastroenterology & hepatology 8(10), 554–564 (2011) [DOI] [PubMed] [Google Scholar]
- 4.Holzheimer, R.G., Mannick, J.A.: Surgical treatment: evidence-based and problem-oriented (2001) [PubMed]
- 5.Tranquillini, C.V., Bernardo, W.M., Brunaldi, V.O., MOURA, E.T.d., Marques, S.B., MOURA, E.G.H.d.: Best polypectomy technique for small and diminutive colorectal polyps: A systematic review and meta-analysis. Arquivos de gastroenterologia 55, 358–368 (2018) [DOI] [PubMed]
- 6.Costamagna, G., Shah, S.K., Riccioni, M.E., Foschia, F., Mutignani, M., Perri, V., Vecchioli, A., Brizi, M.G., Picciocchi, A., Marano, P.: A prospective trial comparing small bowel radiographs and video capsule endoscopy for suspected small bowel disease. Gastroenterology 123(4), 999–1005 (2002) [DOI] [PubMed] [Google Scholar]
- 7.Iddan, G., Meron, G., Glukhovsky, A., Swain, P.: Wireless capsule endoscopy. Nature 405(6785), 417–417 (2000) [DOI] [PubMed] [Google Scholar]
- 8.Omori, T., Hara, T., Sakasai, S., Kambayashi, H., Murasugi, S., Ito, A., Nakamura, S., Tokushige, K.: Does the pillcam sb3 capsule endoscopy system improve image reading efficiency irrespective of experience? a pilot study. Endoscopy international open 6(06), 669–675 (2018) [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Jha, D., Ali, S., Tomar, N.K., Johansen, H.D., Johansen, D., Rittscher, J., Riegler, M.A., Halvorsen, P.: Real-time polyp detection, localization and segmentation in colonoscopy using deep learning. Ieee Access 9, 40496–40510 (2021) [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Urban, G., Tripathi, P., Alkayali, T., Mittal, M., Jalali, F., Karnes, W., Baldi, P.: Deep learning localizes and identifies polyps in real time with 96% accuracy in screening colonoscopy. Gastroenterology 155(4), 1069–1078 (2018) [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Gross, S., Stehle, T., Behrens, A., Auer, R., Aach, T., Winograd, R., Trautwein, C., Tischendorf, J.: A comparison of blood vessel features and local binary patterns for colorectal polyp classification. In: Medical Imaging 2009: Computer-Aided Diagnosis, vol. 7260, pp. 758–765 (2009). SPIE
- 12.Iwahori, Y., Hattori, A., Adachi, Y., Bhuyan, M.K., Woodham, R.J., Kasugai, K.: Automatic detection of polyp using hessian filter and hog features. Procedia computer science 60, 730–739 (2015) [Google Scholar]
- 13.Amber, A., Iwahori, Y., Bhuyan, M.K., Woodham, R.J., Kasugai, K.: Feature point based polyp tracking in endoscopic videos. In: 2015 3rd International Conference on Applied Computing and Information Technology/2nd International Conference on Computational Science and Intelligence, pp. 299–304 (2015). IEEE
- 14.Sasmal, P., Bhuyan, M.K., Iwahori, Y., Kasugai, K.: Colonoscopic polyp classification using local shape and texture features. IEEE Access 9, 92629–92639 (2021) [Google Scholar]
- 15.Pogorelov, K., Ostroukhova, O., Jeppsson, M., Espeland, H., Griwodz, C., de Lange, T., Johansen, D., Riegler, M., Halvorsen, P.: Deep learning and hand-crafted feature based approaches for polyp detection in medical videos. In: 2018 IEEE 31st International Symposium on Computer-Based Medical Systems (CBMS), pp. 381–386 (2018). IEEE
- 16.Hmoud Al-Adhaileh, M., Mohammed Senan, E., Alsaade, W., Aldhyani, T.H.H., Alsharif, N., Abdullah Alqarni, A., Uddin, M.I., Alzahrani, M.Y., Alzain, E.D., Jadhav, M.E.: Deep learning algorithms for detection and classification of gastrointestinal diseases. Complexity 2021, 1–12 (2021)
- 17.Goel, N., Kaur, S., Gunjan, D., Mahapatra, S.: Dilated cnn for abnormality detection in wireless capsule endoscopy images. Soft Computing, 1–17 (2022)
- 18.Jain, S., Seal, A., Ojha, A.: A convolutional neural network with meta-feature learning for wireless capsule endoscopy image classification. Journal of Medical and Biological Engineering 43(4), 475–494 (2023) [Google Scholar]
- 19.Jia, X., Meng, M.Q.-H.: A deep convolutional neural network for bleeding detection in wireless capsule endoscopy images. In: 2016 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC), pp. 639–642 (2016). IEEE [DOI] [PubMed]
- 20.Ronneberger, O., Fischer, P., Brox, T.: U-net: Convolutional networks for biomedical image segmentation. In: Medical Image Computing and Computer-Assisted Intervention–MICCAI 2015: 18th International Conference, Munich, Germany, October 5-9, 2015, Proceedings, Part III 18, pp. 234–241 (2015). Springer
- 21.Badrinarayanan, V., Kendall, A., Cipolla, R.: Segnet: A deep convolutional encoder-decoder architecture for image segmentation. IEEE transactions on pattern analysis and machine intelligence 39(12), 2481–2495 (2017) [DOI] [PubMed] [Google Scholar]
- 22.Dosovitskiy, A., Beyer, L., Kolesnikov, A., Weissenborn, D., Zhai, X., Unterthiner, T., Dehghani, M., Minderer, M., Heigold, G., Gelly, S., et al.: An image is worth 16x16 words: Transformers for image recognition at scale. arXiv preprint arXiv:2010.11929 (2020)
- 23.Jia, X., Meng, M.Q.-H.: Gastrointestinal bleeding detection in wireless capsule endoscopy images using handcrafted and cnn features. In: 2017 39th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC), pp. 3154–3157 (2017). IEEE [DOI] [PubMed]
- 24.Yuan, Y., Li, B., Meng, M.Q.-H.: Bleeding frame and region detection in the wireless capsule endoscopy video. IEEE journal of biomedical and health informatics 20(2), 624–630 (2015) [DOI] [PubMed] [Google Scholar]
- 25.Yuan, Y., Wang, J., Li, B., Meng, M.Q.-H.: Saliency based ulcer detection for wireless capsule endoscopy diagnosis. IEEE transactions on medical imaging 34(10), 2046–2057 (2015) [DOI] [PubMed] [Google Scholar]
- 26.Jain, S., Seal, A., Ojha, A., Krejcar, O., Bureš, J., Tachecí, I., Yazidi, A.: Detection of abnormality in wireless capsule endoscopy images using fractal features. Computers in biology and medicine 127, 104094 (2020) [DOI] [PubMed] [Google Scholar]
- 27.Sánchez-González, A., García-Zapirain, B., Sierra-Sosa, D., Elmaghraby, A.: Automatized colon polyp segmentation via contour region analysis. Computers in biology and medicine 100, 152–164 (2018) [DOI] [PubMed] [Google Scholar]
- 28.Jia, X., Xing, X., Yuan, Y., Xing, L., Meng, M.Q.-H.: Wireless capsule endoscopy: A new tool for cancer screening in the colon with deep-learning-based polyp recognition. Proceedings of the IEEE 108(1), 178–197 (2019) [Google Scholar]
- 29.Shin, Y., Balasingham, I.: Comparison of hand-craft feature based svm and cnn based deep learning framework for automatic polyp classification. In: 2017 39th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC), pp. 3277–3280 (2017). IEEE [DOI] [PubMed]
- 30.Guo, Y., Bernal, J., J. Matuszewski, B.: Polyp segmentation with fully convolutional deep neural networks–extended evaluation study. Journal of Imaging 6(7), 69 (2020) [DOI] [PMC free article] [PubMed]
- 31.Tajbakhsh, N., Gurudu, S.R., Liang, J.: Automated polyp detection in colonoscopy videos using shape and context information. IEEE transactions on medical imaging 35(2), 630–644 (2015) [DOI] [PubMed] [Google Scholar]
- 32.Bernal, J., Sánchez, F.J., Fernández-Esparrach, G., Gil, D., Rodríguez, C., Vilariño, F.: Wm-dova maps for accurate polyp highlighting in colonoscopy: Validation vs. saliency maps from physicians. Computerized medical imaging and graphics 43, 99–111 (2015) [DOI] [PubMed]
- 33.Mahmud, T., Paul, B., Fattah, S.A.: Polypsegnet: A modified encoder-decoder architecture for automated polyp segmentation from colonoscopy images. Computers in Biology and Medicine 128, 104119 (2021) [DOI] [PubMed] [Google Scholar]
- 34.Jha, D., Smedsrud, P.H., Riegler, M.A., Johansen, D., De Lange, T., Halvorsen, P., Johansen, H.D.: Resunet++: An advanced architecture for medical image segmentation. In: 2019 IEEE International Symposium on Multimedia (ISM), pp. 225–2255 (2019). IEEE
- 35.Ta, N., Chen, H., Lyu, Y., Wu, T.: Ble-net: boundary learning and enhancement network for polyp segmentation. Multimedia Systems 29(5), 3041–3054 (2023) [Google Scholar]
- 36.Qadir, H.A., Shin, Y., Solhusvik, J., Bergsland, J., Aabakken, L., Balasingham, I.: Polyp detection and segmentation using mask r-cnn: Does a deeper feature extractor cnn always perform better? In: 2019 13th International Symposium on Medical Information and Communication Technology (ISMICT), pp. 1–6 (2019). IEEE
- 37.Jain, S., Seal, A., Ojha, A., Yazidi, A., Bures, J., Tacheci, I., Krejcar, O.: A deep cnn model for anomaly detection and localization in wireless capsule endoscopy images. Computers in Biology and Medicine 137, 104789 (2021) [DOI] [PubMed] [Google Scholar]
- 38.Lafraxo, S., Souaidi, M., El Ansari, M., Koutti, L.: Semantic segmentation of digestive abnormalities from wce images by using attresu-net architecture. Life 13(3), 719 (2023) [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Oukdach, Y., Kerkaou, Z., El Ansari, M., Koutti, L., Fouad El Ouafdi, A., De Lange, T.: Vitca-net: a framework for disease detection in video capsule endoscopy images using a vision transformer and convolutional neural network with a specific attention mechanism. Multimedia Tools and Applications, 1–20 (2024)
- 40.Bai, L., Wang, L., Chen, T., Zhao, Y., Ren, H.: Transformer-based disease identification for small-scale imbalanced capsule endoscopy dataset. Electronics 11(17), 2747 (2022) [Google Scholar]
- 41.Oukdach, Y., Kerkaou, Z., Ansari, M.E., Koutti, L., Ouafdi, A.F.E.: Conv-vit: Feature fusion-based detection of gastrointestinal abnormalities using cnn and vit in wce images. In: 2023 10th International Conference on Wireless Networks and Mobile Communications (WINCOM), pp. 1–6 (2023). 10.1109/WINCOM59760.2023.10322944
- 42.Hosain, A.S., Islam, M., Mehedi, M.H.K., Kabir, I.E., Khan, Z.T.: Gastrointestinal disorder detection with a transformer based approach. In: 2022 IEEE 13th Annual Information Technology, Electronics and Mobile Communication Conference (IEMCON), pp. 0280–0285 (2022). IEEE
- 43.Chen, J., Lu, Y., Yu, Q., Luo, X., Adeli, E., Wang, Y., Lu, L., Yuille, A.L., Zhou, Y.: Transunet: Transformers make strong encoders for medical image segmentation. arXiv preprint arXiv:2102.04306 (2021)
- 44.Alam, M.J., Fattah, S.A.: Sr-attnet: An interpretable stretch–relax attention based deep neural network for polyp segmentation in colonoscopy images. Computers in Biology and Medicine 160, 106945 (2023) [DOI] [PubMed] [Google Scholar]
- 45.Jha, D., Smedsrud, P.H., Riegler, M.A., Halvorsen, P., de Lange, T., Johansen, D., Johansen, H.D.: Kvasir-seg: A segmented polyp dataset. In: MultiMedia Modeling: 26th International Conference, MMM 2020, Daejeon, South Korea, January 5–8, 2020, Proceedings, Part II 26, pp. 451–462 (2020). Springer
- 46.Silva, J., Histace, A., Romain, O., Dray, X., Granado, B.: Toward embedded detection of polyps in wce images for early diagnosis of colorectal cancer. International journal of computer assisted radiology and surgery 9, 283–293 (2014) [DOI] [PubMed] [Google Scholar]
- 47.Jha, D., Tomar, N.K., Ali, S., Riegler, M.A., Johansen, H.D., Johansen, D., de Lange, T., Halvorsen, P.: Nanonet: Real-time polyp segmentation in video capsule endoscopy and colonoscopy. In: Proceedings of the 2021 IEEE 34th International Symposium on Computer-Based Medical Systems (CBMS), pp. 37–43 (2021)
- 48.LeCun, Y., Bottou, L., Bengio, Y., Haffner, P.: Gradient-based learning applied to document recognition. Proceedings of the IEEE 86(11), 2278–2324 (1998) [Google Scholar]
- 49.Zhou, Z., Rahman Siddiquee, M.M., Tajbakhsh, N., Liang, J.: Unet++: A nested u-net architecture for medical image segmentation. In: Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support: 4th International Workshop, DLMIA 2018, and 8th International Workshop, ML-CDS 2018, Held in Conjunction with MICCAI 2018, Granada, Spain, September 20, 2018, Proceedings 4, pp. 3–11 (2018). Springer [DOI] [PMC free article] [PubMed]
- 50.Xiao, X., Lian, S., Luo, Z., Li, S.: Weighted res-unet for high-quality retina vessel segmentation. In: 2018 9th International Conference on Information Technology in Medicine and Education (ITME), pp. 327–331 (2018). IEEE
- 51.Ibtehaz, N., Rahman, M.S.: Multiresunet: Rethinking the u-net architecture for multimodal biomedical image segmentation. Neural networks 121, 74–87 (2020) [DOI] [PubMed] [Google Scholar]
- 52.Oktay, O., Schlemper, J., Folgoc, L.L., Lee, M., Heinrich, M., Misawa, K., Mori, K., McDonagh, S., Hammerla, N.Y., Kainz, B., et al.: Attention u-net: Learning where to look for the pancreas. arXiv preprint arXiv:1804.03999 (2018)
- 53.Fang, Y., Chen, C., Yuan, Y., Tong, K.-y.: Selective feature aggregation network with area-boundary constraints for polyp segmentation. In: Medical Image Computing and Computer Assisted Intervention–MICCAI 2019: 22nd International Conference, Shenzhen, China, October 13–17, 2019, Proceedings, Part I 22, pp. 302–310 (2019). Springer
- 54.Fan, D.-P., Ji, G.-P., Zhou, T., Chen, G., Fu, H., Shen, J., Shao, L.: Pranet: Parallel reverse attention network for polyp segmentation. In: International Conference on Medical Image Computing and Computer-assisted Intervention, pp. 263–273 (2020). Springer
- 55.Huang, C.-H., Wu, H.-Y., Lin, Y.-L.: Hardnet-mseg: A simple encoder-decoder polyp segmentation neural network that achieves over 0.9 mean dice and 86 fps. arXiv preprint arXiv:2101.07172 (2021)
- 56.Yeung, M., Sala, E., Schönlieb, C.-B., Rundo, L.: Focus u-net: A novel dual attention-gated cnn for polyp segmentation during colonoscopy. Computers in biology and medicine 137, 104815 (2021) [DOI] [PMC free article] [PubMed] [Google Scholar]
3.相关文献解读
胃肠道疾病分割和定位是研究人员的一个活跃领域,已经付出了大量努力来开发息肉分割的支持决策系统[23–25]。尤其是息肉,考虑到医生在识别和定位息肉方面面临的挑战,息肉是早期检测和精确分割的最常见疾病之一。过去提出了各种方法来检测和定位 GI 图像中的息肉区域。首先提出的方法是基于手工制作的描述符的传统技术,以获得纹理和几何特征等低级特征。Iwahori 等 [12] 提出了一种自动化的息肉检测方法,该方法将 Hessian 滤波器与定向梯度直方图 (HOG) 结合使用。Hessian 滤波器用于提取潜在的息肉区域,然后从这些区域提取 HOG 特征。这些功能的选择是使用 Random Forests 和 RealAdaBoost 以自适应方式完成的。最后,他们使用 K-Means++ 方法组合各种息肉区域。他们的研究结果表明,与 RealAdaBoost 相比,Random Forests 实现了高灵敏度和特异性。Jain等[26]使用基于RGBL通道图像分形维数的特征来区分胃肠道内的正常和异常情况。它们显示结果以在两个不同的数据集上进行分类:KID 数据集和私有数据集。在他们的实验中,他们使用了各种机器学习分类器,包括 KNN、SVM、RF 和 Naive Bayesian 网络。在他们的研究中,Sanchez-Gonzalez等[27]从CVC数据集中的图像中提取了边缘及其区域的形状、颜色和曲率等特征。然后,他们利用各种分类器(例如 Naive Bayes、MultiBoost AB 和 AdaBoost)来选择性地选择这些特征。这种方法使他们能够识别息肉受影响区域的边界,并通过连接这些识别的边缘来进行后续分割。然而,息肉数据集中特征的可变性对手工方法在识别息肉区域和准确定位它们方面的性能造成了限制。
基于深度学习的卷积神经网络因其能够自动学习特征而在息肉区域分析中证明了其有效性[28,29]。 Guo 等 [30] 通过将 FCN 网络转换为扩张的 ResFCN 来调整 FCN 网络以进行息肉分割,其中他们结合了残差连接,采用高核大小的扩张卷积,并集成了融合子网络。他们的工作利用了内窥镜视觉GIANA息肉分割挑战数据集,包括CVC-ColonDB [31]和CVC-ClinicDB [32]。在他们的研究 [33] 中,作者提出了一个名为 PolypSegNet 的框架。他们的方法提出了一种基于编码器-解码器的深度学习架构。它结合了深度扩张起始 (DDI) 阻滞来捕获各种感受区域的特征。此外,深度融合跳跃模块 (DFSM) 有效地链接编码器层和解码器层,从而实现上下文信息的传递。此外,深度重建模块 (DRM) 优化了多尺度解码特征图,以改进重建。Jha等[34]介绍了ResUNet++方法,这是一种用于语义分割的神经网络。它集成了残差和挤压块、注意力块和闭孔空间金字塔池 (ASPP)。即使在处理小型图像数据集时,他们的模型也表现出了效率。在他们的研究中,Ta等[35]在边界增强模块中引入了边界感知注意力块。此模块利用编码器功能和部分解码器输出来生成 polyp 掩码。Qadir等[36]利用Mask R-CNN作为特征提取器,采用各种CNN架构,包括ResNet50、ResNet101和InceptionResNetV2,在结肠镜图像中进行息肉区域分割。这种方法在 CVC-ColonDB 数据集上取得了可接受的结果。在 [37] 中,作者介绍了一种深度卷积神经网络,旨在检测 WCE 图像中的异常。最初,他们训练了一个有效的基于注意力的 CNN,将图像分为四类之一:息肉、血管、炎症或正常。随后,采用了 Grad-CAM++ 和自定义 SegNet 的融合来对异常图像中的异常区域进行分割。在他们的研究中,Lafraxo 等人 [38] 介绍了一种名为 AttResU-Net 的端到端架构,它结合了 U-Net 框架内的注意力机制和残差单元。这种集成增强了息肉和出血分割的性能,从而提供了改进的分割结果。另一方面,transformer 对胃肠道异常分析产生了显着影响,尽管它们最初是为自然语言处理任务而设计的 [39]。Dosovitskiy等[22]提出了第一个基于自我注意机制的视觉转换器。[40] 中介绍的工作利用具有空间池配置的 transformer 神经网络来解决 WCE 图像中的二进制和多分类挑战。通过池化的应用,视觉转换器的空间设计表现出增强的性能,小规模胶囊内窥镜数据集的显着改进证明了这一点 [41]。[42] 中的作者介绍了一种依靠视觉转换器来识别来自 WCE 图像的胃肠道疾病。他们进行了比较检查,将基于 transformer 的方法的性能与预先训练的 CNN 模型(特别是 DenseNet201)进行了比较。他们的实验表明,视觉转换器在一系列定量性能评估指标上优于 DenseNet201。Chen等[43]介绍了TransUnet框架,该框架采用基于变压器的网络,具有混合ViT编码器和上采样的CNN解码器。这种混合 ViT 集成了 CNN 和变压器元件。TransUnet 获得的结果强调了在编码器中使用变压器进行息肉分割的有效性。[44] 中的作者介绍了一种称为 SR-AttNet 的深度学习框架,专为分割结直肠息肉而设计。该技术采用编码器-解码器架构,将非扩展和扩展滤波相结合,以捕获来自附近和远距离区域的信息,同时还可以理解图像的深度。为了增强模型,他们在每个空间编码器-解码器之间加入了四重跳跃连接,并实施了 Feature-to-Mask 管道来管理扩展和非扩展特征,以实现最终预测。此外,他们的方法结合了一个名为 SR-Attention 的注意力机制,该机制的灵感来自 Stretch-Relax 概念。这个注意力系统创造了具有显著变化的空间特征,这些特征被用来生成有价值的注意力掩码来选择特征。
4.方法论
在本节中,我们将描述定位现有胃肠道息肉的建议方法,该方法利用受基于 Unet 的编码器-解码器模型启发的 2D 分割架构。编码器采用视觉转换器来捕获全局特征,解码器使用多个基于卷积块的注意力机制从图像中提取局部特征。以下是我们系统的概述。
-
我们构建了一个基于编码器的 Vision Transformer 来获得更高级别的功能。
-
基于解码器的 CNN 用于通过多个跳过连接从解码器中提取不同规模的特征。
-
在解码器内的各个点应用压缩和激发注意力,以增强其对最关键图像区域的关注。
-
为了防止解码器中的梯度消失,我们直接从编码器采用带有自定义注意力机制的残差块来保持基本功能。
以下小节提供了所提议系统的每个组件的具体细节,而说明在 GI 图像中定位息肉的方法的整体示意图如图 2 所示。2.

拟议的结肠镜检查和 WCE 图像中息肉定位系统的整体结构
4.1数据准备
在所提出的系统中,我们使用五个数据集进行了实验:CVC clinicDB [32]、CVC colonDB [31]、kvasir-SEG [45]、ETIS-Larib PolypDB [46] 和 Kvasir Capsule-SEG [47]。每个数据集都表现出独特的特征分布,并且由于图像数量有限,通常需要各种增强。为了应对这一挑战,我们将数据增强技术应用于每个数据集。常见的增强方法,包括翻转、亮度、裁剪、旋转、高斯噪声和缩放,应用于除 Kvasir Capsule-SEG 数据集之外的所有数据集,该数据集仅包含 55 个息肉图像。对于这个特定的数据集,我们对每张图像执行了 20 次增强作,总共产生了 1,100 张具有不同特征的合成图像。图 3 展示了 CVC 诊所、CVC 结肠、Kvasir-SEG 和 Etis Larib 数据集的图像增广示例,而图 3 显示了4 提供了应用于 Kvasir Capsule-SEG 数据集的 20 种增强策略。

一些数据增强应用于 CVC Clinic、CVC Colon、Kvasir-SEG 和 Etis larib 数据集

Representation of the twenty augmentation techniques applied to the Kvasir Capsule-SEG dataset: a Zoom (0.7); b Rotate (20); c Brightness (2.0); d Vertical flip; e Horizontal flip; f Random Crop (256, 256); g Random Noise (0.6); h Translate (20, 20); i Shear (0.2); j Contrast (1.5); k Blur (5); l Sharpen (1.5); m Rotate 90 (1); n Rotate 90 (2); o Scale (0.8); p Scale (1.2); q Gamma Correction (1.5); r Color Shift (20); s Grayscale (1); t Rotated 90° counter-clockwise
4.2U-Net 概述
U-Net 最初由 Ronneberger 等人 [20] 引入,用于生物医学图像分割,作为各种图像分割任务的首选架构而广受欢迎。这种架构植根于全卷积网络的基础 [48]。U-Net 模型包括两个关键组件:编码器和解码器路径。编码器的作用是使用一系列具有 3x3 滤波器的卷积层提取深度特征,然后是 ReLU 激活层和 MaxPooling 层。相反,解码器负责创建输出分割映射,采用上采样、卷积、ReLU 激活和 MaxPooling 层等作。值得注意的是,U-Net 的一个关键方面是集成跳过连接,将编码器的每个阶段链接到解码器中的相应阶段。这些跳过连接在提供基本功能方面发挥着至关重要的作用,最终增强了模型生成越来越准确的分割结果的能力。
4.3Vision Transformer 编码器
从前面提供的 U-net 描述中汲取灵感,我们将 Vision Transformer [22] 集成到我们提议的系统的编码器部分。Vision Transformer 是第一款专为图像分类设计的视觉转换器。它的架构植根于 transformer 编码器中的自注意力机制,该机制最初用于自然语言处理任务。在所提出的系统中,我们从头开始开发了视觉转换器,并将其作为所提出的息肉分割方法的编码器组件。这种集成旨在捕获 WCE 和结肠镜检查图像中存在的总体依赖关系。
ViT 编码器模型旨在处理尺寸为 (H、W、C) 的输入图像,其中 H、W 和 C 分别表示图像的高度、宽度和通道数。然后将此图像划分为 N 个展平的补丁,表示为 with dimensions ,其中每个补丁的大小为 (P, P)。修补程序总数 N 的计算公式为 N = 。将这些补丁转换为具有维度的单个向量 X 后,应用一个密集层将 X 转换为大小为 D 的新向量 E,称为嵌入 E[N, D]。由于为息肉分割任务实施了 ViT,因此未添加类嵌入。为了生成最终的向量 Z,将 1D 位置嵌入引入线性嵌入图像块 E,这对于保持 transformer 中的序列顺序至关重要。随后,将上一步中获得的 Z 向量馈送到 transformer encoder 模块中,以生成上下文向量。变压器编码器由多层多头自注意力 (MHSA) 和 MLP 模块组成。层规范化在每个块之前应用,并且跳过连接在每个块之后存在。MLP 模块由两个全连接层组成,采用高斯误差线性单位 (GELU) 作为非线性的激活函数。跳过连接,其中 i 范围从 1 到 4,用于将特征以各种比例传递给解码器部分。这些跳过连接在促进编码器的高级语义信息与解码器的细粒度细节的融合方面起着至关重要的作用。
4.4基于 CNN 解码器的注意力
所提出的方法中使用的解码器与 U-Net 架构中使用的解码器有相似之处。它由一系列从头开始的卷积层块组成,目的是对从编码器获得的特征集进行重新采样。与 U-Net 网络非常相似,其中不同的特征分辨率与解码器融合在一起,我们合并了一组表示𝑆我为 的跳过连接,范围从 1 到 4。这些连接使我们能够从 transformer 编码器中提取一系列特征,每个特征的大小𝐻×𝑊𝑃2×𝑘为 ,随后重塑为 。𝐻𝑃×𝑊𝑃×𝑘对于每个 resolution 𝑆我,嵌入特征都会转换回输入空间。此转换涉及一个包含 3x3 卷积层的块,然后应用归一化层。堆叠变压器的输出特征通过2×2反卷积层体验到分辨率增加。此外,最终变压器的输出特征也经历了类似的分辨率增强步骤,通过应用2×2反卷积层并将结果与前一个变压器的输出连接起来来实现。然后,这些融合的特征通过3×3卷积层,并通过2×2反卷积层进行上采样。整个过程对所有连续的层都是重复的,一直延伸到初始输入分辨率。最后,结果通过1×1具有 softmax 激活函数的卷积层进行处理,该层生成输出掩码预测。
为了增强解码器强调基本图像区域的能力,我们在解码器架构的各个点集成了挤压和激发注意力机制。如图 1 所示。5、SE 机制是一个两步过程。最初,它通过全局平均池化将特征图压缩到单个通道中,并在空间维度上平均值。随后,采用紧凑的神经网络模块来获取特定于通道的权重集合。该模块将压缩的特征图作为输入,并应用非线性变换来生成权重集。

说明挤压和激发的注意力机制的示意图
此外,如图 1 所示。6 和 7 中,我们在残余块中加入了双注意机制,以确保保留从最后一个变压器获得的有价值的特征。这有双重目的:防止梯度消失问题,并促进将最关键的特征传输到解码器网络中的最终卷积层。DAM 采用简化且高效的流程,该流程结合了像素级和通道级注意力机制。如图 1 所示。7,像素级注意力机制由两个连续的卷积层组成,一个是 3x3 的 28 个滤波器,另一个是 1 个滤波器。另一方面,通道注意力机制包含三个卷积层,每个卷积层分别为 3x3 和 128、64 和 1 个滤波器。DAM 的输出与残差块的输入功能集成在一起,对应于编码器部分中最后一个变压器的输出。DAM 特征是按照 Eqs 中描述的方程获得的。1、2、3、4 和 5。索引 (i, j) 表示特征图矩阵中的位置,而 m, n 表示卷积核中的空间位置,是输入特征图 W 中位置 的像素值。, , 是位置 (m, n) 处卷积运算的权重,b 表示偏差项。这一战略步骤起着至关重要的作用,特别是考虑到小尺寸息肉的普遍存在。利用基于残差块的注意力机制,使模型能够有效地保留高级和低级特征,从而为所提出的系统的整体成功做出贡献。


包含双重注意机制的残差块的可视化表示

残差块内使用的 dual attention 机制的结构
5结果与讨论
本节深入介绍了用于息肉分割的数据集、实施细节以及进行的各种实验。此外,它还介绍了所提出模型的定量和定性结果,将它们与胃肠道息肉定位的最新最新方法进行了比较。
5.1数据集和实施详细信息
所提出的系统在其实验中利用了五个数据集:CVC clinicDB [32]、CVC colonDB [31]、kvasir-SEG [45]、ETIS-Larib PolypDB [46]和Kvasir Capsule-SEG [47]。所有这些数据集都是公开可用的,每个数据集都有其独特的特征分布。表 1 显示了有关每个数据集的一些详细信息。如 “数据准备” 部分所述,我们将数据增强技术应用于训练集,以生成额外的合成息肉图像。为此,已使用离线数据增强策略。为了降低计算要求,所有图像的大小都调整为 ,并且每个数据集都以 80:20 的比例拆分,以便进行训练和测试。
表 1.数据增强技术前后每个数据集中包含息肉的图像数量
| 数据 | 增强前 | 增强后 | 图像大小 |
|---|---|---|---|
| CVC-ClinicDB | 612 | 2712 | 384 × 288 |
| Kvasir-SEG | 1000 | 4800 | 626 × 546 |
| CVC-结肠 | 300 | 1344 | 574 × 500 |
| ETIS-拉里布 | 196 | 1020 | 1256 × 966 |
| Kvasir Capsule-SEG | 55 | 1100 | 336 × 336 |
拟议的系统对所有数据集进行训练和评估,将每个数据集用于训练目的,其余数据集用于测试。使用具有最新 TensorFlow 版本的 Keras 作为后端进行实施,在具有 64 GB RAM 和 24 GB NVIDIA GeForce RTX 3090 GPU 的 Intel i7 处理器上运行。训练过程包括处理 100 个 epoch 的 RGB 图像,使用学习率为 0.001 的 Adam 优化器,并集成三个损失函数:分类交叉熵 (BCE)、骰子损失 (Dice) 和 BCE+Dice。此外,还存在提前停止机制,当验证损失在 15 个 epoch 后停滞时,学习率会降低 0.01 倍,以缓解过拟合问题。表 2 列出了所提出的编码器-解码器系统的整个超参数。为了评估所提出的方法的性能,我们利用了分割任务的常用指标,包括平均骰子系数 (mDc)、平均交并比 (mIoU)、精度 (P)、召回率 (R) 和 -score。以下方程显示了每个指标的数学公式,以及本研究中考虑的损失函数。术语 TP、TN、FP 和 FN 分别表示 True Positive、True Negative、False Positive 和 False Negative。此处,y 和 分别表示真实输出和预测输出。

表 2.用于训练所提出的用于息肉定位的编码器-解码器的超参数
| 超参数 | 值 |
|---|---|
| 图像大小 | 256×256 |
| 补丁大小 | 16×16 |
| 投影尺寸 (k) | 64 |
| 刀头数量 | 8 |
| 变压器单元 | [128, 64] |
| 变压器层 | 13 |
| 批量大小 | 16 |
| 优化 | 亚当 |
| 学习率 | 0.001 |
| 权重衰减率 | 0.0001 |
| 纪元数 | 100 |
5.2性能评估和分析
5.2.1定量结果
所提出的系统具有类似于 Unet 模型的架构,利用编码器和解码器中的 ConvNets 层来提取和重新采样特征。所提出的方法结合了卷积运算提取局部特征的潜力与变压器从图像中学习全局信息的变革能力。编码器采用 ViT 来捕获高级特征和重要区域,利用多头注意力机制。同时,特征重采样和分布是通过基于注意力的卷积块实现的。我们从 Eqs 中定义的三个损失函数的实验开始我们的分析。11、12 和 13。使用这些损失函数的目的是量化所提出的模型生成的预测像素掩码与真实像素掩码之间的差异。从本质上讲,损失函数用于衡量预测值和实际值之间的误差。通过评估不同的损失函数,每个损失函数都封装了预测误差的不同方面,我们的目标是辨别哪一个更适合我们的特定任务。如表 3 所示,综合损失 (BCE + Dice) 在所有数据集的几乎所有指标中始终优于所有指标,但 CVC 诊所数据集的召回率和𝐹β=2评分除外。值得注意的是,BCE 损失函数在 CVC 诊所数据集上获得了最高的召回率和𝐹β=2分数,而实施相同的损失函数在 Etis Larib 数据集上产生了 Dice 系数和𝐹β=2分数的最佳结果。当训练将 Dice 作为损失函数时,该模型在 CVC 结肠和 Etis Larib 数据集上都表现出色。此外,还强调了优化过程中综合损失的有效性,在所有指标上都显示了有效的结果,在某些情况下只有细微的差异。该实验证实了它能够最大限度地减少真实像素掩码和预测像素掩码之间的误差。因此,我们选择它作为其余实验的主要损失函数。
表 3.The comparison results of using three different loss functions in the training of the proposed model
| Datasets | Loss | P | R | mIoU | mDc | 𝑭𝜷=𝟐 |
|---|---|---|---|---|---|---|
| CVC Clinic | BCE | 0.8989 | 0.9106 | 0.8956 | 0.9046 | 0.9082 |
| Dice | 0.9006 | 0.9049 | 0.8917 | 0.9025 | 0.9039 | |
| BCE+Dice | 0.9038 | 0.9081 | 0.8981 | 0.9059 | 0.9072 | |
| CVC colon | BCE | 0.9042 | 0.9180 | 0.9016 | 0.9107 | 0.9150 |
| Dice | 0.9047 | 0.9182 | 0.9020 | 0.9109 | 0.9151 | |
| BCE+Dice | 0.9072 | 0.9178 | 0.9042 | 0.9121 | 0.9154 | |
| Kvasir-SEG | BCE | 0.7916 | 0.8135 | 0.7724 | 0.8001 | 0.8073 |
| Dice | 0.8001 | 0.8177 | 0.7850 | 0.8064 | 0.8104 | |
| BCE+Dice | 0.8144 | 0.8211 | 0.8012 | 0.8230 | 0.8352 | |
| Etis Larib | BCE | 0.9155 | 0.9101 | 0.9046 | 0.9175 | 0.9140 |
| Dice | 0.9161 | 0.9174 | 0.9025 | 0.9016 | 0.9051 | |
| BCE+Dice | 0.9196 | 0.9145 | 0.9131 | 0.9118 | 0.9134 | |
| Kvasir Capsule-SEG | BCE | 0.4829 | 0.5278 | 0.4661 | 0.5018 | 0.5161 |
| Dice | 0.4919 | 0.5301 | 0.4703 | 0.5089 | 0.5133 | |
| BCE+Dice | 0.5009 | 0.5418 | 0.4912 | 0.5122 | 0.5372 |
为了评估模型的性能和所提出的系统中每个元素的贡献,我们采用了一项称为消融研究的实验来辨别每个组件对所提出的 UViT-Seg 对息肉定位性能的影响。该实验涉及通过删除和添加挤压注意力机制和基于残差的双重注意力块来评估所提出的系统。表 4 显示了该实验获得的结果,表明所提出的方法通过组合其所有组成部分来获得最佳结果。在 CVC Clinic 数据集上,所提出的模型在考虑单个元素时展示了细微的结果。单独使用注意力机制并排除解码器中的残差块,可产生 89% 的高精度。相反,当孤立使用残差块时,mIoU、mDc 和 显示更高的值,并且忽略了 SE 关注。对于 Kvasir-SEG 和 CVC Colon 数据集,在所提出的模型中专门使用残差块展示了其优于仅依赖注意力的优势。当采用 SE 注意力时,mDc 和 在 Etis Larib 数据集上分别表现出 89.23% 和 89.98% 的高值。同时,mIoU 、 precision 和 recall 通过对残差块的独占集成,分别以 89.66%、88.67% 和 89.26% 的值获得第二个峰值结果。在本实验中使用的最后一个数据集 Kvasir Capsule-SEG 中,当系统仅使用残差块构建时,精度和 mDc 展示了强大的二级性能。相比之下,mIoU、recall 并通过独家结合 attention 表现出高水平的性能。在排除 SE 关注和残差块的情况下获得最低的结果。然而,消融实验的结果最终表明,将这两个元素整合到所提出的系统中可以获得在 GI 图像中定位息肉的最佳模型性能。对五个数据集进行了额外的实验,涉及在残差块内有和没有所提出的双重注意力机制的模型评估。本实验的目的是评估在残差块中加入双注意力机制对模型性能的影响。该实验的结果如表 5 所示,生动地证明了残差块内双重注意力机制的潜力。将 DAM 集成到残差数据块中可产生卓越的结果,在所有数据集和指标(CVC Colon 数据集除外)中实现高性能。在这种特殊情况下,残差块中没有 DAM 的版本表现出 91.82% 的显着召回率。尽管如此,整体实验还是坚实地验证了 DAM 模块的有效性。它在帮助残差块捕获最关键的区域方面发挥着关键作用,这些区域来自位于跳过连接 S4 之后的最后一个反卷积层处理的重采样特征。
Table 4.
The outcomes of the ablation experiments conducted on the proposed model across the five datasets
| Datasets | SE mechanism | Resdual block | P | R | mIoU | mDc | 𝑭𝜷=𝟐 |
|---|---|---|---|---|---|---|---|
| CVC Clinic | × | × | 0.8648 | 0.8724 | 0.8537 | 0.8684 | 0.8607 |
| ✓ | × | 0.8900 | 0.8959 | 0.8715 | 0.8959 | 0.8909 | |
| × | ✓ | 0.8828 | 0.8976 | 0.8868 | 0.8900 | 0.8985 | |
| ✓ | ✓ | 0.9038 | 0.9081 | 0.8981 | 0.9059 | 0.9072 | |
| Kvasir-SEG | × | × | 0.7647 | 0.7993 | 0.7417 | 0.7836 | 0.7973 |
| ✓ | × | 0.7874 | 0.7583 | 0.7148 | 0.7651 | 0.7595 | |
| × | ✓ | 0.7837 | 0.8026 | 0.7731 | 0.8005 | 0.8127 | |
| ✓ | ✓ | 0.8144 | 0.8211 | 0.8012 | 0.8230 | 0.8352 | |
| CVC Colon | × | × | 0.8817 | 0.8833 | 0.8606 | 0.8790 | 0.8871 |
| ✓ | × | 0.8847 | 0.8943 | 0.8765 | 0.8877 | 0.8916 | |
| × | ✓ | 0.8872 | 0.8907 | 0.8787 | 0.8975 | 0.8991 | |
| ✓ | ✓ | 0.9072 | 0.9178 | 0.9042 | 0.9121 | 0.9154 | |
| Etis Larib | × | × | 0.8857 | 0.8739 | 0.8891 | 0.8847 | 0.8742 |
| ✓ | × | 0.8901 | 0.8954 | 0.8891 | 0.8923 | 0.8998 | |
| × | ✓ | 0.8966 | 0.8867 | 0.8926 | 0.8861 | 0.8964 | |
| ✓ | ✓ | 0.9196 | 0.9145 | 0.9131 | 0.9118 | 0.9134 | |
| Kvasir Capsule-SEG | × | × | 0.4878 | 0.4710 | 0.4313 | 0.4736 | 0.4803 |
| ✓ | × | 0.4419 | 0.5090 | 0.4651 | 0.4826 | 0.5132 | |
| × | ✓ | 0.4928 | 0.4999 | 0.4528 | 0.4939 | 0.4966 | |
| ✓ | ✓ | 0.5009 | 0.5418 | 0.4912 | 0.5122 | 0.5372 |
Table 5.
Effect of Dual Attention mechanism within the residual blocks on the proposed model performance, where the bold values indicate the superior results
| Residual Block | P | R | mIoU | 𝐹𝛽=2 | mDc |
|---|---|---|---|---|---|
| CVC Clinic | |||||
| With DAM | 0.9038 | 0.9081 | 0.8981 | 0.9059 | 0.9072 |
| Without DAM | 0.8868 | 0.8906 | 0.8835 | 0.8935 | 0.8977 |
| CVC Colon | |||||
| With DAM | 0.9072 | 0.9178 | 0.9042 | 0.9121 | 0.9154 |
| Without DAM | 0.8996 | 0.9182 | 0.8969 | 0.8980 | 0.9038 |
| Kvasir-SEG | |||||
| With DAM | 0.8144 | 0.8211 | 0.8012 | 0.8230 | 0.8352 |
| Without DAM | 0.7731 | 0.8243 | 0.7648 | 0.7957 | 0.8111 |
| Etis Larib | |||||
| With DAM | 0.9196 | 0.9145 | 0.9131 | 0.9118 | 0.9134 |
| Without DAM | 0.8903 | 0.9091 | 0.8984 | 0.9046 | 0.9073 |
| Kvasir Capsule-SEG | |||||
| With DAM | 0.5009 | 0.5418 | 0.4912 | 0.5122 | 0.5372 |
| Without DAM | 0.4692 | 0.5154 | 0.4469 | 0.4893 | 0.5038 |
为了评估训练模型的泛化性能,对五个数据集进行了一组全面的实验,每个数据集专门用于测试在另一个数据集上训练的模型。这种全面的方法旨在评估模型在所用数据集中的表现,以及它是否可以有效地概括其学习的模式。表 6 显示了该实验的综合结果,揭示了性能指标的有见地的趋势和变化。当在一个数据集上训练并在其他数据集(不包括 Kvasir Capsule-SEG 数据集)上进行测试时,该模型表现出强大的分割能力。特别是,在测试期间,CVC Clinic 数据集的训练在 CVC Colon、Kvasir-SEG 和 Etis Larib 数据集上表现出值得称赞的性能,凸显了其多功能性。同样,在 CVC Colon 上训练的模型展示了倒数有效性,在 CVC Clinic、Kvasir-SEG 和 Etis Larib 数据集上产生了值得注意的结果。在 Kvasir-SEG 和 Etis Larib 数据集上进行训练也证明了可接受的泛化性能。在 Kvasir-SEG 作为训练数据集场景的更广泛背景下,分割模型在多个数据集中表现出值得称道的泛化,展示了稳健的性能指标。值得注意的是,当应用于 CVC Clinic 数据集时,该模型分别实现了令人印象深刻的 87.27%、90.51%、86.43%、88.76% 和 89.77% 的准确率、召回率、借据𝐹β=2、分数和骰子系数值。这一趋势在其他数据集中继续存在,CVC 结肠数据集的大量结果证明了这一点,其中精确率、召回率、mIoU、𝐹β=2分数和 mDc 达到 87.70%、91.09%、86.73%、89.15% 和 90.23%,证明了该模型的一致有效性。此外,Etis Larib 数据集还展示了该模型的熟练程度,记录了 91.98%、89.85%、87.77%、90.67% 和 90.13% 的精确率、召回率、IoU、𝐹β=2分数和 mDc。这些发现强调了该模型在适应各种数据集方面的多功能性,并突出了其在不同数据特征中保持高分割性能的能力。然而,在训练和测试期间,Kvasir Capsule-SEG 数据集出现了一个例外,将较低的结果归因于其有限的大小(55 张原始图像)和增强策略的潜在影响。此外,与其他数据集中以小息肉为主的不同,Kvasir Capsule-SEG 的原始图像中存在大息肉,这可能是造成这种性能差异的原因。
Table 6.
The generalization performance of the proposed UViT-Seg, where each dataset is used for training and the others for evaluation
| ↓ Evaluation Data/Metrics → | P | R | mIoU | 𝐹𝛽=2 | mDc |
|---|---|---|---|---|---|
| CVC clinic: Train | |||||
| CVC Colon | 0.8692 | 0.9042 | 0.8542 | 0.8837 | 0.8951 |
| Kvasir-SEG | 0.7374 | 0.8015 | 0.7107 | 0.7653 | 0.7844 |
| Etis Larib | 0.8915 | 0.8877 | 0.8486 | 0.8888 | 0.8879 |
| Kvasir Capsule-SEG | 0.3624 | 0.5405 | 0.3566 | 0.4250 | 0.4817 |
| CVC colon: Train | |||||
| CVC Clinic | 0.8617 | 0.9106 | 0.8584 | 0.8843 | 0.8996 |
| Kvasir-SEG | 0.7271 | 0.8314 | 0.7255 | 0.7744 | 0.8050 |
| Etis Larib | 0.8967 | 0.9372 | 0.8931 | 0.9159 | 0.9284 |
| Kvasir Capsule-SEG | 0.4003 | 0.5502 | 0.3987 | 0.4507 | 0.4980 |
| Kvasir-SEG: Train | |||||
| CVC clinic | 0.8727 | 0.9051 | 0.8643 | 0.8876 | 0.8977 |
| CVC Colon | 0.8770 | 0.9109 | 0.8673 | 0.8915 | 0.9023 |
| Etis Larib | 0.9198 | 0.8985 | 0.8777 | 0.9067 | 0.9013 |
| Kvasir Capsule-SEG | 0.4395 | 0.5488 | 0.4368 | 0.4814 | 0.5164 |
| Etis Larib: Train | |||||
| CVC clinic | 0.8679 | 0.8365 | 0.7944 | 0.8475 | 0.8399 |
| CVC Colon | 0.8642 | 0.9044 | 0.8492 | 0.8810 | 0.8940 |
| Kvasir-SEG | 0.7444 | 0.8043 | 0.7180 | 0.7691 | 0.7880 |
| Kvasir Capsule-SEG | 0.3528 | 0.5461 | 0.3485 | 0.4205 | 0.4822 |
| Kvasir Capsule-SEG: Train | |||||
| CVC Clinic | 0.8776 | 0.7990 | 0.7681 | 0.8324 | 0.8112 |
| CVC Colon | 0.8900 | 0.7798 | 0.7572 | 0.8290 | 0.7983 |
| Kvasir-SEG | 0.7865 | 0.6895 | 0.6504 | 0.7261 | 0.7020 |
| Etis Larib | 0.9166 | 0.7445 | 0.7291 | 0.8137 | 0.7695 |
Table 7.UViT-Seg 与常见医学成像分割方法的比较分析。粗体值表示最佳结果,而蓝色值表示次佳结果
| ↓ Models/Metrics → | P | R | mIoU | 𝐹𝛽=2 | mDc |
|---|---|---|---|---|---|
| CVC Clinic | |||||
| Unet [20] | 0.8080 | 0.8120 | 0.7961 | 0.8048 | 0.7991 |
| Unet ++ [49] | 0.7882 | 0.8011 | 0.7456 | 0.7773 | 0.7869 |
| ResUnet [50] | 0.8408 | 0.8640 | 0.8408 | 0.8746 | 0.8574 |
| MultiResUnet [51] | 0.9048 | 0.8711 | 0.9019 | 0.8879 | 0.8798 |
| AttentionUnet [52] | 0.8214 | 0.8349 | 0.8125 | 0.7630 | 0.7841 |
| The proposed Model | 0.9038 | 0.9081 | 0.8981 | 0.9059 | 0.9072 |
| CVC Colon | |||||
| Unet | 0.8976 | 0.9181 | 0.8952 | 0.9071 | 0.9135 |
| Unet ++ | 0.8594 | 0.9210 | 0.8594 | 0.8868 | 0.9062 |
| ResUnet | 0.8594 | 0.9210 | 0.8594 | 0.8868 | 0.9062 |
| MultiResUnet | 0.9014 | 0.9192 | 0.8999 | 0.9098 | 0.9153 |
| AttentionUnet | 0.8917 | 0.9197 | 0.8907 | 0.9043 | 0.9131 |
| The proposed Model | 0.9072 | 0.9178 | 0.9042 | 0.9121 | 0.9154 |
| Kvasir-SEG | |||||
| Unet | 0.7867 | 0.8275 | 0.7810 | 0.8048 | 0.8173 |
| Unet ++ | 0.7163 | 0.8335 | 0.7162 | 0.7678 | 0.8038 |
| ResUnet | 0.7721 | 0.8201 | 0.7598 | 0.7934 | 0.8083 |
| MultiResUnet | 0.8042 | 0.8264 | 0.7971 | 0.8144 | 0.8210 |
| AttentionUnet | 0.7990 | 0.8210 | 0.7868 | 0.8088 | 0.8155 |
| The proposed Model | 0.8144 | 0.8211 | 0.8012 | 0.8230 | 0.8352 |
| Etis Larib | |||||
| Unet | 0.9268 | 0.9391 | 0.9048 | 0.9028 | 0.9065 |
| Unet ++ | 0.8893 | 0.9411 | 0.8893 | 0.8936 | 0.8997 |
| ResUnet | 0.9173 | 0.9370 | 0.8934 | 0.9067 | 0.9028 |
| MultiResUnet | 0.9310 | 0.9401 | 0.9000 | 0.9102 | 0.9118 |
| AttentionUnet | 0.9295 | 0.9396 | 0.9081 | 0.9045 | 0.9075 |
| The proposed Model | 0.9196 | 0.9145 | 0.9131 | 0.9118 | 0.9134 |
| Kvasir Capsule-SEG | |||||
| Unet | 0.4805 | 0.5439 | 0.4748 | 0.5088 | 0.5284 |
| Unet ++ | 0.4657 | 0.4266 | 0.3678 | 0.4364 | 0.4283 |
| ResUnet | 0.4602 | 0.5310 | 0.4465 | 0.4918 | 0.5142 |
| MultiResUnet | 0.5010 | 0.5356 | 0.4866 | 0.5159 | 0.5268 |
| AttentionUnet | 0.4637 | 0.5243 | 0.4438 | 0.4898 | 0.5090 |
| The proposed Model | 0.5009 | 0.5418 | 0.4912 | 0.5122 | 0.5372 |
Table 8.五种常见医学成像方法和 5 重验证的 UViT-Seg 的分割性能
| Datasets | CVC | CVC | Kvasir | Etis | Kvasir | |||||
|---|---|---|---|---|---|---|---|---|---|---|
| Clinic | Colon | SEG | Larib | Capsule-SEG | ||||||
| Models | mDc | mIoU | mDc | mIoU | Dc | mIoU | mDc | mIoU | mDc | mIoU |
| [20] | 0.890 | 0.869 | 0.870 | 0.851 | 0.801 | 0.774 | 0.903 | 0.890 | 0.450 | 0.388 |
| [49] | 0.874 | 0.840 | 0.863 | 0.820 | 0.765 | 0.713 | 0.885 | 0.850 | 0.425 | 0.357 |
| [50] | 0.877 | 0.846 | 0.832 | 0.800 | 0.756 | 0.701 | 0.900 | 0.881 | 0.436 | 0.371 |
| [51] | 0.896 | 0.901 | 0.893 | 0.872 | 0.805 | 0.782 | 0.891 | 0.864 | 0.474 | 0.422 |
| [52] | 0.889 | 0.867 | 0.876 | 0.844 | 0.797 | 0.768 | 0.908 | 0.881 | 0.430 | 0.365 |
| UViT-Seg | 0.886 | 0.860 | 0.876 | 0.859 | 0.727 | 0.655 | 0.914 | 0.890 | 0.411 | 0.336 |
Table 9.使用 Wilcoxon 秩和检验对所提出的 UViT-Seg 与常见的最先进方法进行统计分析。以粗体突出显示的值显示 p 值≥0.05
| Dataset | CVC | CVC | Kvasir | Etis | Kvasir | |||||
|---|---|---|---|---|---|---|---|---|---|---|
| Clinic | Colon | SEG | Larib | Capsule-SEG | ||||||
| P-v | P-v | P-v | P-v | P-v | P-v | P-v | P-v | P-v | P-v | |
| (Dc) | (IoU) | (Dc) | (IoU) | (Dc) | (IoU) | (Dc) | (IoU) | (Dc) | (IoU) | |
| A | 0.009 | 0.073 | 0.007 | 0.007 | 0.001 | 0.004 | 0.007 | 0.081 | 0.070 | 0.078 |
| B | 0.051 | 0.032 | 0.009 | 0.009 | 0.002 | 0.006 | 0.006 | 0.004 | 0.084 | 0.076 |
| C | 0.061 | 0.048 | 0.009 | 0.009 | 0.004 | 0.004 | 0.042 | 0.063 | 0.075 | 0.068 |
| D | 0.018 | 0.005 | 0.004 | 0.043 | 0.017 | 0.033 | 0.009 | 0.008 | 0.073 | 0.076 |
| E | 0.003 | 0.041 | 0.005 | 0.011 | 0.032 | 0.037 | 0.046 | 0.030 | 0.022 | 0.001 |
Table 10.UViT-Seg 与最新息肉分割方法的比较研究结果
| CVC ColonDB | CVC Clinic | Kvasir-SEG | Etis LaribDB | |||||
|---|---|---|---|---|---|---|---|---|
| Models | mDc | mIoU | mDc | mIoU | mDc | mIoU | mDc | mIoU |
| SFA [53] | 0.469 | 0.347 | 0.700 | 0.607 | 0.723 | 0.611 | 0.297 | 0.217 |
| PraNet [54] | 0.709 | 0.640 | 0.899 | 0.849 | 0.898 | 0.840 | 0.628 | 0.567 |
| HarDNet-MSEG [55] | 0.731 | 0.660 | 0.932 | 0.882 | 0.912 | 0.857 | 0.677 | 0.613 |
| Focus U-Net [56] | 0.878 | 0.804 | 0.938 | 0.889 | 0.910 | 0.853 | 0.832 | 0.757 |
| SR-AttNet [44] | 0.665 | 0.539 | 0.786 | 0.693 | 0.871 | 0.806 | 0.476 | 0.355 |
| UViT-Seg | 0.915 | 0.902 | 0.907 | 0.898 | 0.835 | 0.801 | 0.913 | 0.9131 |
该模型的复杂性会影响其在实际场景中的部署,因为临床服务不仅需要高准确性,还需要效率和可解释性。在医学图像分割的背景下,例如我们研究中考虑的任务,部署高度复杂的模型可能会在临床环境中的实际实施中带来挑战。表 11 显示了所提出的方法与其他息肉分割方法之间的复杂性分析。与其他方法相比,所提出的 UViT-Seg 脱颖而出,因为它需要更少的计算资源并显着减少训练时间。
Table 11.
Trainable parameters of the proposed approach and other approaches
| Models | Trainable Parameters (M) |
|---|---|
| Unet | 31.04 |
| Unet++ | 9.04 |
| ResUnet | 8.22 |
| Multi-ResUnet | 7.24 |
| Attention Unet | 8.13 |
| ParaNet | 32.55 |
| HarDNet-MSEG | 33.34 |
| SR-AttNet | 31.1 |
| UViT-Seg | 2.24 |
5.3定性结果
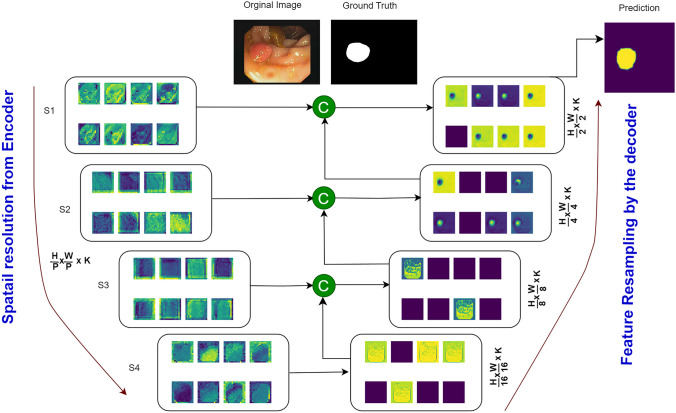
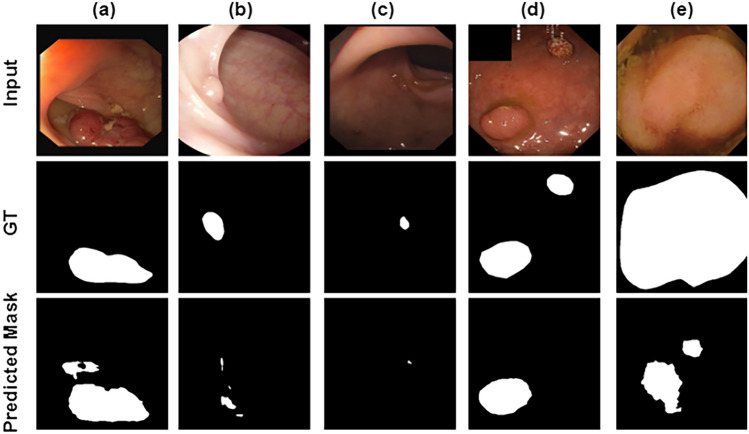
在本节中,我们深入探讨了所提出的用于息肉定位的 UViT-Seg 的可视化性能。我们的探索从可视化编码器和解码器每个阶段的功能开始。图 8 提供了在每个 skip connection 处获得的特征的可视化表示,其中 i .每组中的特征保持一致的分辨率,并被馈送到解码器中,以促进各个阶段的特征重建。每个输出通过反卷积层进行重采样,然后通过一组卷积层,从而增强特征捕获。然后,生成的特征将与 的下一个输出融合。检查图 .8 中,我们观察到特征连接过程使模型能够在每个阶段系统地捕获基本特征。事实证明,这种特征的顺序捕获对于在不同层面获得有趣的见解至关重要,有助于模型在从最后一层生成最终预测之前有效地定位息肉区域。在详细检查了每个阶段的特征并彻底理解了模型的解释,尤其是它对复杂细节的强调之后,我们直观地展示了模型在图 5 中不同训练测试分片的五个不同数据集上的性能。9. 此图展示了原始图像、地面实况 (GT)、预测掩码和 Grad-CAM 可视化。我们研究中考虑的数据集显示出息肉特征的显着变化,包括质地、形状和大小的差异(从小息肉到大息肉)。该模型在识别每个测试数据集中的大多数息肉方面表现出值得称道的熟练程度,无论它们的大小如何。这种稳健的性能表明该模型对不同息肉特征的适应性,使其成为在各种条件下进行一致息肉检测的多功能工具。此外,Grad-CAM 分析为模型的决策过程提供了更多见解,突出了其专注于包含息肉的区域的能力。这种可视化效果不仅可以验证模型的预测,还有助于了解对其准确性影响最大的特征。通过 Grad-CAM 成功识别关键区域强调了该模型在 GI 图像中精确定位息肉的潜力。
图 8.使用提出的 UViT-Seg 在各种分辨率下可视化预测特征,测试图像来自 CVC 诊所数据集

图.9由 UViT-Seg 生成的预测掩码的可视化表示以及使用 Grad CAM 识别的突出显示区域
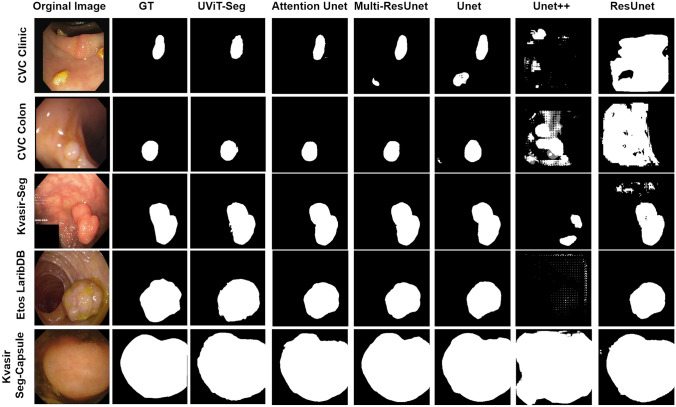
每个智能系统面临的主要挑战在于它在具有不同特征分布的新数据上的性能。为了应对这一挑战,我们提出了所提出的 UViT-Seg 的额外视觉表示,以验证其泛化能力。该实验需要在一个数据集上训练模型,然后评估其在其他数据集上的性能。我们的目标是评估该模型对息肉不同情况的适应性,包括大小、形状和一系列其他特征的变化。具体来说,这项工作中使用的数据集都显示出自己独特的特征。例如,Kvasir Capsule-SEG 数据集包括在特定条件下使用 WCE 技术捕获的图像,其中大多数息肉都很大。相比之下,其他数据集是用消化管捕获的,导致完全不同的特征,尤其是在它们表示小尺寸息肉方面。然而,图 .图10介绍了该实验的可视化结果以及模型在显示小息肉和中位息肉的数据集(CVC Clinic、CVC colon、Etis Larib 和 Kvasir-SEG)上的泛化能力。即使它们的特征存在显着差异,该模型在大多数情况下也表现出识别和定位息肉区域的显着能力。可视化分析表明,在一个数据集上训练的模型成功地将其性能扩展到具有不同小息肉和中位息肉的数据集,这表明具有很有前途的泛化水平。如前所述,Kvasir Capsule-SEG 数据集的情况是独一无二的。它仅包括 55 个原始息肉及其相应的面具,所有这些息肉都很大。这些特定条件可能会影响模型的泛化性能。如图 1 所示。10,当此数据集用于测试时,预测的息肉在大多数情况下看起来较小或中等大小,除了使用 Kvasir-SEG 训练模型的情况,预测的息肉保持其较大的形状。同样,当用于训练时,预测的息肉始终以较大的大小显示。这种差异引发了关于数据集的特征如何影响模型在不同息肉大小之间泛化的能力的考虑。尽管 Kvasir Capsule-SEG 带来了巨大的挑战,但 UViT-Seg 在某些情况下表现出预测息肉区域的能力。在其他情况下,它可以熟练地识别大小不同的息肉区域。相反,在处理其他数据集时,该模型证明了其泛化能力,展示了其适应性,这是息肉在形态和大小方面表现出相当大的多样性的真实场景的关键属性。此外,我们还将所提出的模型与几种常见的医学图像分割方法进行了比较分析,包括 Unet、Unet++、ResUnet、AttentionUnet 和 Mult-ResUnet。目的是评估所提出的模型与其他已建立的医学分割方法相比的性能。图 11 提供了比较方法的可视化表示,揭示了所提出的模型在所有评估数据集中准确定位息肉方面始终如一地取得出色的性能。AttentionUnet 还表现出定位形状略有变化的息肉区域的能力。然而,Unet 和 Multi-ResUnet 显示出一些混淆,尤其是在 CVC Clinic 和 CVC Colon 数据集的情况下。值得注意的是,Unet++ 和 ResUnet 模型在有效分割息肉方面遇到了挑战并表现出重大困难,但 Etis Larib 和 Kvasir Capsule-SEG 等情况除外,在这些情况下,ResUnet 模型成功识别了息肉区域。

图10.可视化 UViT-Seg 在所有数据集上的泛化性能,其中每个数据集用于训练,而其他数据集用于评估
图11.可视化 UViT-Seg 和其他常见医学图像分割方法的性能
然而,虽然所提出的 UViT-Seg 模型证明了它在 GI 图像中的息肉分割中的有效性,但它也表现出一定的局限性。在图 .12 中,我们展示了与拟议的 UViT-Seg 相关的故障案例的可视化表示。在我们的实验中,我们确定了预测失败的三个主要原因。首先,在许多情况下,与包含息肉的区域相比,法线区域显示出非常相似的特征,这给模型区分它们带来了重大挑战。其次,所提出的方法可能难以准确定位位于边界不明确的深部区域的息肉。最后,在某些情况下,某些图像中的正常区域与其他图像中的息肉区域共享特征,这可能会影响 UViT-Seg 的预测性能。为了解决这些限制,必须持续努力。结合其他数据增强技术有望提高模型区分特征的能力。此外,准备更广泛的数据集,特别是关注不同的息肉,特别是较大的息肉,可能会减轻与 Kvasir Capsule-SEG 中预测失败相关的挑战,并增强泛化过程。
图12.与建议的 UViT-Seg 相关的故障工况的可视化表示。来自每个数据集的示例图像
6.结论与代码实现
息肉分割在医学领域提出了重大挑战,主要是由于与息肉特征相关的各种复杂性,例如颜色、质地、形状以及与胃肠道筛查技术相关的其他因素。深度学习模型的最新进展因其与生俱来的有效提取特征的能力而展示了卓越的性能。这些模型中的大多数采用采用卷积层的编码器-解码器架构,这在学习 GI 图像中息肉区域的不同全局表示时可能不够健壮。在这项研究中,我们介绍了 UViT-Seg,这是一个专为结肠镜检查和 WCE 图像中的自动息肉分割而设计的框架。与大多数基于 CNN 的医学图像分割方法不同,尤其是在息肉中,UViT-Seg 的显着特点在于它能够从图像中提取特征表示。这是通过在编码器中部署视觉转换器来实现的,捕获高级特征以更深入地了解息肉区域。同时,解码器模块使用一组基于 squeeze 和 excitation 机制的反卷积和卷积块捕获低级特征。此外,与残差块集成的双注意力机制增强了模型保留输入 GI 图像基本特征的能力。实验结果来自用于训练和测试的五个不同的数据集,每个数据集都有独特的特征分布和不同的息肉条件。结果证实了 UViT-Seg 能够精确定位图像中的息肉。此外,UViT-Seg 利用一个数据集进行训练,其余四个数据集进行评估,验证其泛化和适应看不见的数据中特征新变化的能力。与五种常见医学分割方法的性能比较证明了 UViT-Seg 的有效性。此外,与最近的几种息肉分割方法相比,所提出的 UViT-Seg 表现出最佳结果,以其复杂性效率而著称。它需要更少的计算资源进行训练和测试,使其更适合实际部署场景。
尽管如此,虽然 UViT-Seg 模型产生了有效的结果,但它仍然遇到了一定的局限性。一个挑战在于它能够区分正常区域和包含息肉的区域,因为这些区域的特征分布非常相似。此外,它很难准确定位深部区域的息肉,尤其是在边界不明确的情况下。此外,由于使用了五个不同的数据集,每个数据集都有其独特的特征,该模型在推广其发现方面面临困难,尤其是在处理较大息肉的定位时。通过解决这些挑战并在未来的工作中对模型进行增强,我们的目标是在 WCE 和结肠镜检查图像中实现更准确的息肉区域定位和识别。这种方法对于边界不明确和尺寸较大的息肉尤其重要。此外,这些改进将推动息肉检测综合系统的开发,使其更能够适应 GI 图像中息肉区域存在的多样化特征分布。
6.1系统功能结构图
6.2系统界面



结尾彩蛋
联系看往期文章末尾点击这里
代码附录
import os
#os.environ["TF_CPP_MIN_LOG_LEVEL"] = "2"
#os.environ["CUDA_VISIBLE_DEVICES"]="0"
import numpy as np
import cv2
from glob import glob
import tensorflow as tf
from tensorflow.keras.metrics import Precision, Recall, MeanIoU
from tensorflow.keras.optimizers import Adam, Nadam, SGD
from tensorflow.keras.callbacks import EarlyStopping, ModelCheckpoint, ReduceLROnPlateau, CSVLogger, TensorBoard
from tensorflow.keras.utils import multi_gpu_model
from sklearn.utils import shuffle
from unet import Unet
from resunet import ResUnet
from m_resunet import ResUnetPlusPlus
from metrics import *
from tf_data import *
from sgdr import *
def shuffling(x, y):
x, y = shuffle(x, y, random_state=42)
return x, y
if __name__ == "__main__":
tf.random.set_seed(42)
np.random.seed(42)
## Path
file_path = "files/"
## Create files folder
try:
os.mkdir("files")
except:
pass
train_path = "new_data/train/"
valid_path = "new_data/valid/"
## Training
train_image_paths = sorted(glob(os.path.join(train_path, "image", "*.jpg")))
train_mask_paths = sorted(glob(os.path.join(train_path, "mask", "*.jpg")))
## Shuffling
train_image_paths, train_mask_paths = shuffling(train_image_paths, train_mask_paths)
## Validation
valid_image_paths = sorted(glob(os.path.join(valid_path, "image", "*.jpg")))
valid_mask_paths = sorted(glob(os.path.join(valid_path, "mask", "*.jpg")))
## Parameters
image_size = 256
batch_size = 16
lr = 1e-5
epochs = 300
model_path = "files/resunetplusplus.h5"
train_dataset = tf_dataset(train_image_paths, train_mask_paths)
valid_dataset = tf_dataset(valid_image_paths, valid_mask_paths)
try:
arch = ResUnetPlusPlus(input_size=image_size)
model = arch.build_model()
model = tf.distribute.MirroredStrategy(model, 4, cpu_merge=False)
print("Training using multiple GPUs..")
except:
arch = ResUnetPlusPlus(input_size=image_size)
model = arch.build_model()
print("Training using single GPU or CPU..")
optimizer = Nadam(learning_rate=lr)
metrics = [dice_coef, MeanIoU(num_classes=2), Recall(), Precision()]
model.compile(loss="binary_crossentropy", optimizer=optimizer, metrics=metrics)
model.summary()
schedule = SGDRScheduler(min_lr=1e-6,
max_lr=1e-2,
steps_per_epoch=np.ceil(epochs/batch_size),
lr_decay=0.9,
cycle_length=5,
mult_factor=1.5)
callbacks = [
ModelCheckpoint(model_path),
# ReduceLROnPlateau(monitor='val_loss', factor=0.1, patience=6),
CSVLogger("files/data.csv"),
TensorBoard(),
EarlyStopping(monitor='val_loss', patience=50, restore_best_weights=False),
schedule
]
train_steps = (len(train_image_paths)//batch_size)
valid_steps = (len(valid_image_paths)//batch_size)
if len(train_image_paths) % batch_size != 0:
train_steps += 1
if len(valid_image_paths) % batch_size != 0:
valid_steps += 1
model.fit(train_dataset,
epochs=epochs,
validation_data=valid_dataset,
steps_per_epoch=train_steps,
validation_steps=valid_steps,
callbacks=callbacks,
shuffle=False)
"""
UNet architecture in Keras TensorFlow
"""
import os
import numpy as np
import cv2
import tensorflow as tf
from tensorflow.keras.layers import *
from tensorflow.keras.models import Model
class Unet:
def __init__(self, input_size=256):
self.input_size = input_size
def build_model(self):
def conv_block(x, n_filter, pool=True):
x = Conv2D(n_filter, (3, 3), padding="same")(x)
x = BatchNormalization()(x)
x = Activation("relu")(x)
x = Conv2D(n_filter, (3, 3), padding="same")(x)
x = BatchNormalization()(x)
x = Activation("relu")(x)
c = x
if pool == True:
x = MaxPooling2D((2, 2), (2, 2))(x)
return c, x
else:
return c
n_filters = [16, 32, 64, 128, 256]
inputs = Input((self.input_size, self.input_size, 3))
c0 = inputs
## Encoder
c1, p1 = conv_block(c0, n_filters[0])
c2, p2 = conv_block(p1, n_filters[1])
c3, p3 = conv_block(p2, n_filters[2])
c4, p4 = conv_block(p3, n_filters[3])
## Bridge
b1 = conv_block(p4, n_filters[4], pool=False)
b2 = conv_block(b1, n_filters[4], pool=False)
## Decoder
d1 = Conv2DTranspose(n_filters[3], (3, 3), padding="same", strides=(2, 2))(b2)
d1 = Concatenate()([d1, c4])
d1 = conv_block(d1, n_filters[3], pool=False)
d2 = Conv2DTranspose(n_filters[3], (3, 3), padding="same", strides=(2, 2))(d1)
d2 = Concatenate()([d2, c3])
d2 = conv_block(d2, n_filters[2], pool=False)
d3 = Conv2DTranspose(n_filters[3], (3, 3), padding="same", strides=(2, 2))(d2)
d3 = Concatenate()([d3, c2])
d3 = conv_block(d3, n_filters[1], pool=False)
d4 = Conv2DTranspose(n_filters[3], (3, 3), padding="same", strides=(2, 2))(d3)
d4 = Concatenate()([d4, c1])
d4 = conv_block(d4, n_filters[0], pool=False)
## output
outputs = Conv2D(1, (1, 1), padding="same")(d4)
outputs = BatchNormalization()(outputs)
outputs = Activation("sigmoid")(outputs)
## Model
model = Model(inputs, outputs)
return model
篇幅有限代码给出部分核心代码
import os
import random
import numpy as np
import cv2
from tqdm import tqdm
from glob import glob
import tifffile as tif
from sklearn.model_selection import train_test_split
from utils import *
from albumentations import (
PadIfNeeded,
HorizontalFlip,
VerticalFlip,
CenterCrop,
Crop,
Compose,
Transpose,
RandomRotate90,
ElasticTransform,
GridDistortion,
OpticalDistortion,
RandomSizedCrop,
OneOf,
CLAHE,
RandomBrightnessContrast,
RandomGamma,
HueSaturationValue,
RGBShift,
RandomBrightness,
RandomContrast,
MotionBlur,
MedianBlur,
GaussianBlur,
GaussNoise,
ChannelShuffle,
CoarseDropout
)
def augment_data(images, masks, save_path, augment=True):
""" Performing data augmentation. """
crop_size = (256, 256)
size = (256, 256)
for image, mask in tqdm(zip(images, masks), total=len(images)):
image_name = image.split("/")[-1].split(".")[0]
mask_name = mask.split("/")[-1].split(".")[0]
x, y = read_data(image, mask)
h, w, c = x.shape
if augment == True:
## Center Crop
aug = CenterCrop(p=1, height=crop_size[0], width=crop_size[0])
augmented = aug(image=x, mask=y)
x1 = augmented['image']
y1 = augmented['mask']
## Crop
x_min = 0
y_min = 0
x_max = x_min + size[1]
y_max = y_min + size[0]
aug = Crop(p=1, x_min=x_min, x_max=x_max, y_min=y_min, y_max=y_max)
augmented = aug(image=x, mask=y)
x2 = augmented['image']
y2 = augmented['mask']
## Random Rotate 90 degree
aug = RandomRotate90(p=1)
augmented = aug(image=x, mask=y)
x3 = augmented['image']
y3 = augmented['mask']
## Transpose
aug = Transpose(p=1)
augmented = aug(image=x, mask=y)
x4 = augmented['image']
y4 = augmented['mask']
## ElasticTransform
aug = ElasticTransform(p=1, alpha=120, sigma=120 * 0.05, alpha_affine=120 * 0.03)
augmented = aug(image=x, mask=y)
x5 = augmented['image']
y5 = augmented['mask']
## Grid Distortion
aug = GridDistortion(p=1)
augmented = aug(image=x, mask=y)
x6 = augmented['image']
y6 = augmented['mask']
## Optical Distortion
aug = OpticalDistortion(p=1, distort_limit=2, shift_limit=0.5)
augmented = aug(image=x, mask=y)
x7 = augmented['image']
y7 = augmented['mask']
## Vertical Flip
aug = VerticalFlip(p=1)
augmented = aug(image=x, mask=y)
x8 = augmented['image']
y8 = augmented['mask']
## Horizontal Flip
aug = HorizontalFlip(p=1)
augmented = aug(image=x, mask=y)
x9 = augmented['image']
y9 = augmented['mask']
## Grayscale
x10 = cv2.cvtColor(x, cv2.COLOR_RGB2GRAY)
y10 = y
## Grayscale Vertical Flip
aug = VerticalFlip(p=1)
augmented = aug(image=x10, mask=y10)
x11 = augmented['image']
y11 = augmented['mask']
## Grayscale Horizontal Flip
aug = HorizontalFlip(p=1)
augmented = aug(image=x10, mask=y10)
x12 = augmented['image']
y12 = augmented['mask']
## Grayscale Center Crop
aug = CenterCrop(p=1, height=crop_size[0], width=crop_size[0])
augmented = aug(image=x10, mask=y10)
x13 = augmented['image']
y13 = augmented['mask']
##
aug = RandomBrightnessContrast(p=1)
augmented = aug(image=x, mask=y)
x14 = augmented['image']
y14 = augmented['mask']
aug = RandomGamma(p=1)
augmented = aug(image=x, mask=y)
x15 = augmented['image']
y15 = augmented['mask']
aug = HueSaturationValue(p=1)
augmented = aug(image=x, mask=y)
x16 = augmented['image']
y16 = augmented['mask']
aug = RGBShift(p=1)
augmented = aug(image=x, mask=y)
x17 = augmented['image']
y17 = augmented['mask']
aug = RandomBrightness(p=1)
augmented = aug(image=x, mask=y)
x18 = augmented['image']
y18 = augmented['mask']
aug = RandomContrast(p=1)
augmented = aug(image=x, mask=y)
x19 = augmented['image']
y19 = augmented['mask']
aug = MotionBlur(p=1, blur_limit=7)
augmented = aug(image=x, mask=y)
x20 = augmented['image']
y20 = augmented['mask']
aug = MedianBlur(p=1, blur_limit=10)
augmented = aug(image=x, mask=y)
x21 = augmented['image']
y21 = augmented['mask']
aug = GaussianBlur(p=1, blur_limit=10)
augmented = aug(image=x, mask=y)
x22 = augmented['image']
y22 = augmented['mask']
aug = GaussNoise(p=1)
augmented = aug(image=x, mask=y)
x23 = augmented['image']
y23 = augmented['mask']
aug = ChannelShuffle(p=1)
augmented = aug(image=x, mask=y)
x24 = augmented['image']
y24 = augmented['mask']
aug = CoarseDropout(p=1, max_holes=8, max_height=32, max_width=32)
augmented = aug(image=x, mask=y)
x25 = augmented['image']
y25 = augmented['mask']
images = [
x, x1, x2, x3, x4, x5, x6, x7, x8, x9, x10,
x11, x12, x13, x14, x15, x16, x17, x18, x19, x20,
x21, x22, x23, x24, x25
]
masks = [
y, y1, y2, y3, y4, y5, y6, y7, y8, y9, y10,
y11, y12, y13, y14, y15, y16, y17, y18, y19, y20,
y21, y22, y23, y24, y25
]
else:
images = [x]
masks = [y]
idx = 0
for i, m in zip(images, masks):
i = cv2.resize(i, size)
m = cv2.resize(m, size)
tmp_image_name = f"{image_name}_{idx}.jpg"
tmp_mask_name = f"{mask_name}_{idx}.jpg"
image_path = os.path.join(save_path, "image/", tmp_image_name)
mask_path = os.path.join(save_path, "mask/", tmp_mask_name)
cv2.imwrite(image_path, i)
cv2.imwrite(mask_path, m)
idx += 1
def load_data(path, split=0.1):
""" Load all the data and then split them into train and valid dataset. """
img_path = glob(os.path.join(path, "images/*"))
msk_path = glob(os.path.join(path, "masks/*"))
train_x, valid_x = train_test_split(img_path, test_size=split, random_state=42)
train_y, valid_y = train_test_split(msk_path, test_size=split, random_state=42)
return (train_x, train_y), (valid_x, valid_y)
def main():
np.random.seed(42)
path = "../../../ml_dataset/Kvasir-SEG/"
(train_x, train_y), (valid_x, valid_y) = load_data(path, split=0.2)
create_dir("new_data/train/image/")
create_dir("new_data/train/mask/")
create_dir("new_data/valid/image/")
create_dir("new_data/valid/mask/")
augment_data(train_x, train_y, "new_data/train/", augment=True)
augment_data(valid_x, valid_y, "new_data/valid/", augment=False)
if __name__ == "__main__":
main()
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容







所有评论(0)